Benutzer:Binse/Baustelle
Neubearbeitung zum Artikel Nukleotide
Nukleotide
[Bearbeiten | Quelltext bearbeiten]Nukleotide im engeren Sinn sind vor allem die Einzelglieder der Kettenmoleküle Desoxyribonukleinsäure, DNA, und Ribonukleinsäure, RNA, deren Namen Nukleinsäuren daran erinnert, dass sie im Zellkern lebender Organismen entdeckt wurden. Indem die DNA vermöge des genetischen Codes Information speichert und die RNA (unter anderem) solche Information übermittelt, ermöglichen sie eine Grundfunktion des Phänomens Leben: die identische Reduplikation. Nur vier Nukleotide als Bausteine der DNA, die in prinzipiell beliebiger Reihenfolge verknüpft sein können, representieren so die gesamte Formenfülle des Lebens. In der RNA findet man dieselben vier Bausteine in leicht modifizierter Form. Ungeachtet ihres Namens erfüllen die Nukleinsäuren die gleichen Funktionen auch in Bakterien und Archäen, die bekanntlich keinen Zellkern besitzen. Außerdem haben auch Eukarioten einen Großteil der Nukleotide außerhalb des Zellkerns als rRNA in den Ribosomen, wo die Proteinsynthese stattfindet. In der Transfer-RNA, deren Funktion es ist, die jeweils vom Code bezeichneten Aminosäuren für die Proteinsynthese heranzuschaffen, finden sich an bestimmten Stellen weitere Nukleotide, dies übrigens unterschiedlich je nach Spezies. Über weitere Funktionen der sehr vielseitigen RNA siehe dort. In Theorien zur Entstehung des Lebens wird RNA gegenüber der DNA als ursprünglicher angesehen, siehe RNA-Welt-Hypothese. Auch Viren, nicht eigentlich lebend aber eine Randerscheinung des Lebens, speichern ihre Information vermittels DNA oder RNA.
Selbständig, als Mononukleotide, spielen sie wichtige und sogar Schlüsselrollen in der Chemie der Zelle, siehe z.B. ATP, gern als die Energiewährung der Zelle bezeichnet, sowie cAMP und cGMP, sogenannte Second Messenger, die der Signalübermittlung in der Zelle dienen. Ein Protein-GTP-Komplex ist notwendig für einen Transportmechanisnus durch die Kernmembran, siehe Ran-GTP, Membrantransport.
Als Halbzeug für die Synthese der Nukleinsäuren hält die Zelle ebenfalls Mononukleotide bereit; für die RNA jederzeit, für die DNA in der Vorbereitung der Mitose. Schließlich sind noch einige Dinukleotide mit unterschiedlichen Funktionen bekannt.
Aufbau
[Bearbeiten | Quelltext bearbeiten]Ein Nukleotid besteht aus drei Komponenten. Dabei ist eine
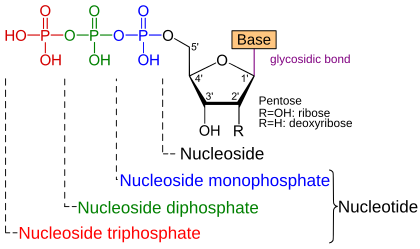
verknüpft mit einer
- Pentose, d.i. ein Zucker mit fünf Kohlenstoffatomen,
sowie mit
- einer Kette von ein bis drei Phosphatgruppen PO4H2
am 5'-Kohlenstoff der Pentose, wobei pro Bindung ein Molekül H2O abgespalten wird. Die Base ist am 1'-Kohlenstoff der Pentose gebunden.
Die Verbindung von Nukleobase und Pentose allein heißt Nukleosid. Demnach ist ein Nukleotid ein Nukleosidphosphat, genauer: ein -mono-, -di- oder -triphosphat.
Alle Verbindungen nach diesem Bauplan sind Nukleotide im weiteren Sinn. Sie bilden eine viel umfangreichere Menge als die aus der Natur bekannten Nukleotide, zumal es keine verbindliche Liste von Nukleobasen zu geben scheint. Dieser Artikel geht nur auf die natürlich vorkommenden Nukleotide ein.
Nukleobasen
[Bearbeiten | Quelltext bearbeiten]Die in der DNA auftretenden Nukleobasen (DNA-Basen) sind Adenin, Guanin, Cytosin und Thymin, kurz A, G, C, T geschrieben. Davon treten A, G. C ebenso in der RNA auf, nur Thymin ist durch das nahe verwandte Uracil (U) ersetzt: Thymin ist ein Methyluracil. Eine plausible Erklärung für diesen Unterschied von DNA und RNA gibt der Artikel Uracil.
 |
 |
 |
 |
 |
 |

| ||||||
| Adenin | Guanin | Cytosin | Uracil | Thymin | 9H-Purin | Pyrimidin |
Für die Regel der Basenpaarung (s.u.) sind T und U äquivalent: Während stets G mit C paart, paart A mit T oder U. Nach ihren Grundgerüsten werden Adenin und Guanin als Purinbasen, salopp Purine, bezeichnet, dagegen Cytosin, Thymin und Uracil als Pyrimidinbasen, salopp Pyrimidine. Die Anbindung an die Pentose geschieht durch Abspaltung von H2O aus der sekundären Aminogruppe (d.i. NH) der Base, die in den Formeln jeweils unten steht, und der 1'-OH-Gruppe des Zuckers, was eine sogenannte N-glycosidische C-N Bindung ergibt. Eine Anzahl von weiteren Nukleobasen finden sich in den schon erwähnten zusätzlichen Nukleotiden bzw. Nukleosiden der tRNAs, s.d. So enthält z.B. das seltene Nukleosid Inosin das Purinderivat Hypoxanthin als Nukleobase.
Zucker und Phosphat
[Bearbeiten | Quelltext bearbeiten]Die Pentose der natürlichen Nukleotide ist Ribose oder Desoxyribose. Wenn die RNA-Welt-Hypothese zutrifft, wofür vieles spricht, hat die Evolution erst mit der ‚Erfindung‘ der DNA das Tor zu der großartigen Vielfalt der lebenden Organismen aufgestoßen. Die mit Desoxyribose statt Ribose gebildete DNA ist nämlich chemisch, insbesondere gegenüber Hydrolyse, sehr viel stabiler als RNA. Mit DNA als korrosionsfestem Speichermedium wurden sehr viel größere Genome möglich, und nur große und mit geringer Fehlerrate kopierbare Genome erlauben die Entwicklung komplexer Organismen und definierter Arten. Die übrigen zellulären Funktionen erfüllt dabei weiterhin die flexiblere vielseitige RNA.
Die Bausteine von RNA und DNA sind einfach phosphatierte Nukleoside. Mononukleotide dagegen spielen auch als Di- und Triphosphate wichtige Rollen im Leben der Zelle.
Nomenklatur
[Bearbeiten | Quelltext bearbeiten]Die Nukleoside der RNA sind mit Ribose und überwiegend mit einer der Basen
- Adenin, Guanin, Cytosin und Uracil
gebildet. Sie tragen eigene Namen:
Daneben gibt es in mRNA regelmäßig das Pseudouridin, das aus Uridin durch eine Umlagerung entsteht. Auch damit ist die Liste nicht vollständig, aber weitere RNA-Nukleoside sind selten.
Die entsprechenden Nukleoside der DNA sind mit Desoxyribose statt Ribose und an Stelle von Uracil mit Thymin gebildet und heißen analog
Diese Liste ist, soweit bekannt, für frisch synthetisierte DNA vollständig. Im weiteren Verlauf wird allerdings Cytosin häufig zu Methylcytosin und weiter zu Hydroxymethylcytosin modifiziert.
Wo man übrigens die Bezeichnung Thymidin findet, die eigentlich die Verbindung von Thymin mit Ribose bezeichnen sollte, ist gewöhnlich Desoxythymidin gemeint. Als Nichtstandardnukleosid kommt die Verbindung von Thymidin und Ribose tatsächlich in bestimmten tRNAs vor. Sie wird dann Ribothymidin oder Methyluridin genannt, was Verwechslungen vermeidet.
Die natürlichen Nukleotide sind dann die Monophosphate, Diphosphate oder Triphosphate der genannten Nukleoside. Ihre Namen werden üblicherweise abgekürzt, wie z.B. ATP, UDP, dTMP.
Das System ist wie folgt: Die Nukleoside werden mit dem Kennbuchstaben A, G, C, T, U ihrer Base bezeichnet (Pseudouridin mit ψ), mit vorgesetztem klein d für Desoxynukleoside. Der Anhang MP, DP oder TP steht für -mono-, -di- oder -triphosphat.
Die obigen Beispiele demnach: ATP ist Adenosintriphosphat, UDP ist Uridindiphosphat und dTMP Desoxythymidinmonophosphat.
Verkettung
[Bearbeiten | Quelltext bearbeiten]
Die Kettenmoleküle DNA und RNA werden gebildet, indem die Phosphatgruppe eines Nukleotids (dort an der 5'-Position des Zuckers gebunden) eine Esterbindung mit der 3'-OH-Gruppe eines weiteren Nukleotids eingeht. In der Zelle wird diese Reaktion durch eines von mehreren Enzymen namens Polymerase vermittelt. Das Nukleotid an einem Kettenende, 5'-Ende genannt, trägt dann am 5'C einen PO4H2-Rest. Der 3'-Kohlenstoff am anderen Kettenende (dem 3'-Ende) die unveränderte OH-Gruppe. Durchläuft man das Molekül vom 5'- zum 3'-Ende, kurz: in 5'-3'-Richtung, so ist jeweils das 3'C-Atom des einen Zuckers mit dem 5'C des nächsten durch eine PO4H-Gruppe verbunden. Die Ketten können sich nach demselben Muster auch zu Ringen schließen. Im einfachsten Fall besteht der Ring aus einem einzigen Nukleotid als 3'-5'-Phosphorsäure-Diester, siehe cAMP und cGMP. Da jeweils ein Molekül Wasser abgespalten wird, handelt es sich beim Verketten um eine Kondensationsreaktion. Die bekannten Polymerasen lassen die Kette stets am 3'-Ende wachsen: Syntheserichtung von Nukleinsäuren ist 5'-3'.
Basenpaarung und genetischer Code
[Bearbeiten | Quelltext bearbeiten]Die Kettenmoleküle DNA und RNA neigen dazu, sich der Länge nach zu einem Molekülpaar zusammenzulagern, wobei die Basen sich paarweise aneinander passen und die Zuckerphosphate diese Paare in zwei Strängen begleiten. Dabei werden keine kovalenten Bindungen geknüpft und eine stabile Konfiguration ergibt sich nur, wenn
erstens
- die beiden Ketten entgegengesetzt orientiert sind, also ein 3'-5'-Strang neben einem 5'-3'-Strang liegt
und zweitens
- die einander gegenüberstehenden Basen räumlich und chemisch zueinander passen.
Da Purine mehr Raum beanspruchen als Pyrimidine, bewirkt die Geometrie, dass stets nur ein Purin mit einem Pyrimidin ‚paart‘, nicht aber Purin mit Purin oder Pyrimidin mit Pyrimidin. Die chemische Passung beruht auf der Bildung von Wasserstoffbrücken und bewirkt im Verein mit der Geometrie, dass nur
- Paarungsregel: Adenin mit Thymin (bzw. Uracil) und Guanin mit Cytosin, kurz: A mit T/U und G mit C
paaren. Basen, Nukleoside oder Nukleotide, die gemäß dieser Regel miteinander paaren, werden zueinander komplementär genannt. DNA- oder RNA-Stränge heißen komplementär, wenn sich bei gegenläufiger Orientierung stets komplementäre Basen gegenüberstehen. In diesem Fall entsteht ein stabiler Doppelstrang.
Die Paarungsregel ist die Grundlage der zellulären Informationsverarbeitung. Der Informationsspeicher, die genetische Substanz der Chromosomen von Pro- wie von Eukarioten besteht aus Paaren komplementärer DNA-Einzelstränge, die sich zu der von Watson und Crick entdekten Doppelhelix umeinanderwinden. Durch die Verdoppelung ist nicht nur die genetische Substanz chemisch und mechanisch stabilisiert, sondern auch die in der Basenfolge enthaltene Information gespiegelt, also doppelt vorhanden. Jeder Strang ist Backup für den anderen.
Bei der Neusynthese von DNA in der Replikation und von RNA bei der Transkription (s.u.) wird jeweils zu einer DNA-Vorlage ein komplementärer Strang aus Mononukleotiden hergestellt. Folglich wird stets auch die vorliegende Information (gespiegelt) kopiert. Das gilt entsprechend bei der reversen Transkription von RNA nach DNA bei Retroviren. Da, wie gesagt, der neue Strang in 5'-3'-Richtung wächst, ist die Leserichtung an der Vorlage stets 3'-5'.
RNA ist in der Konformation vielseitiger als DNA. Eine RNA-Doppelhelix bildet das Genom gewisser Viren. Die für die Proteinsynthese benötigten tRNAs der Zellen anderseits sind einzelsträngig und falten sich so, dass komplementäre Partien miteinender paaren und dazwischen ungepaarte Bereiche als Schleifen stehen bleiben. In den zwei Untereinheiten der Ribosomen schließlich sind RNA-Einzelstränge mit Proteinmolekülen zu stabilen Komplexen verwoben.
Ein Großteil der genetischen Information einer Zelle besteht in den ‚Bauplänen‘ seiner Proteine. Da ein Protein ein Kettenmolekül aus Aminosäuren ist, genügt als Bauplan die Aufzählung derselben. Diese Information speichert und nutzt die Zelle als eine Folge von Tripeln von Nukleotiden (von denen wir nur die Basen anschreiben): ATG bzw. AUG bedeutet Methionin, ACC Threonin, GGG Glycin, um nur drei Beispiele zu nennen. Die Tripel heißen in diesem Zusammenhang Codons. Von den 43=64 Codons wirken drei als Stoppsignal. Da jedes der übrigen 61 Codons tatsächlich eine von den 20 zur Auswahl stehenden Aminosäuren bezeichnet (und auch verwendet wird), codieren meist mehrere Codons für eine Aminosäure: Der genetische Code ist, wie man sagt, entartet. Die Tatsache, dass dabei in 8 von 16 Fällen bereits die ersten zwei Basen eines Codons die Aminosäure eindeutig bestimmen, führt zu der Vermutung, die dritte Position habe ursprünglich nur der Interpunktion gedient, so dass in diesem frühen Zustand der Code effektiv nur aus 16 Paaren bestand und lebende Materie höchstens soviele Aminosäuren enthielt. Über die Mechanismen, mit denen von der DNA zunächst eine RNA-Abschrift (mRNA) genommen und diese in ein Protein übersetzt wird, siehe die Artikel Transkription und Translation.
Die Stichworte Doppelhelix, Basenpaarung, genetischer Code, Transkription/Translation und Replikation umreißen zweifellos eines der wichtigsten Forschungsergebnisse des zwanzigsten Jahrhunderts: die im Prinzip vollständige Erklärung des Phänomens Leben aus Sicht der Chemie.
Energietransport
[Bearbeiten | Quelltext bearbeiten]Die Triphosphate der Nukleoside sind energiereicher als die Diphosphate und diese wiederum energiereicher als die Monophosphate. Für die Synthese von DNA oder RNA werden die benötigten Nukleotide als Triphosphate von der Polymerase angenommen obwohl sie als Monophosphate eingebaut werden. Die Energiedifferenz treibt die Gesamtreaktion. Bei vielen Reaktionen, die auf den ersten Blick nichts mit Adenosin zu tun haben, ist es doch beteiligt, indem es als ATP in den Prozess ein, und als AMP aus ihm austritt, und so die für die Reaktion benötigte Energie liefert. Für die ‚Aufladung‘, Phosphorylierung, von AMP oder ADP zu ATP gibt es verschiedene Wege. Bei aeroben, d.h. Sauerstoff atmenden eukaryotischen Zellen schaffen dies vor allem die Mitochondrien im Wege der Atmungskette; hierzu s.a. Substratkettenphosphorylierung sowie Creatinphosphat. Andere Nukleoside können auf dem Umweg über ATP aufgeladen werden nach dem Schema XMP+ATP → XDP+AMP, XDP+ATP → XTP+AMP, so etwa für X=U.
Methylierung
[Bearbeiten | Quelltext bearbeiten]Während frisch synthetisierte DNA nur die oben genannten vier Standard-Desoxynukleotide enthält, werden später viele Cytosine, und zwar solche aus 5'CG3'-Sequenzen (CG-Dinukleotiden) zu Methylcytosinen modifiziert. Das geschieht meist an allen CG-Dinukleotiden einer ganzen Region und zwar an beiden Strängen, da die beteiligten Enzyme die Stränge gar nicht unterscheiden können. Hinsichtlich der Paarungsregel reagiert Methylcytosin wie Cytosin. Die methylierten Regionen der DNA werden aber in einem komplizierten Prozess, der auch die beteiligten Histone modifiziert, fester an ihre Nukleosomen gebunden, was das Ablesen (die Transcription) betroffener Gene behindert oder sogar ganz blockiert. Diesen Mechanismus benutzt der Organismus mit dem folgenden Trick, um Genaktivitäten über die Zellteilung hinweg zu regulieren. Einer methylierten 5'CG3' Sequenz auf einem Strang liegt nach der Paarungsregel 3'GC5' gegenüber, das ist aber dasselbe wie 5'CG3' und folglich auch methyliert. Bei der Replikation, bei der jedem Strang ein neuer Gegenstrang synthetisiert wird, wird die Methylierung zunächst ignoriert. Jeder der zwei Doppelstränge enthält nun (alte) methylierte gepaart mit (neuen) nichtmethylierten CG-Dinukleotiden. Anschliessend sucht ein spezielles Enzyme nach solchen halbmethylierten Stellen der Doppelhelix und methyliert auch die andere Seite. So wird das Methylierungsmuster von der Mutter- auf die Tochterzellen übertragen. Durch demethylierende Enzyme kann der Organismus die Aktivität der betroffenen Gene auch wieder anschalten.
Mechanismen, die die Genaktivität über die Zellteilung hinweg beeinflussen, ohne dabei die Basenfolge des genetischen Materials zu verändern, fasst man unter dem Begriff Epigenetik zusammen. Hierzu siehe auch Genomische Prägung, DNA-Methylierung, CpG-Insel, Differenzierung.
Literatur
[Bearbeiten | Quelltext bearbeiten]- J. M. Berg, J. L. Tymoczko, L. Stryer: Biochemie. 6. Auflage. Spektrum Akademischer Verlag, Elsevier GmbH, München 2007; ISBN 978-3-8274-1800-5
- Rolf Knippers: Molekulare Genetik. 9. komplett überarbeitete Auflage, Thieme Verlag 2006, ISBN 3-13-477009-1
- Bruce Alberts u. a.: Molekularbiologie der Zelle. 4. Auflage. Wiley-VCH, Weinheim 2003, ISBN 3-527-30492-4